CoVGAS - コロナウイルスゲノム配列解析 専用ソフト
CoVGAS
販売とサポート終了のお知らせ
Nextclade GISAID や Pangolin の状況変化などを始めとし、様々な環境が変化しているためにCovGAS製品の販売とサポートを終了いたします。
現在お使いのソフトウエアは以下の条件で継続してお使いいただけます。(データベースの自動更新は行えません)
CPU:Core i5/ i7以上(8世代以上を推奨)
メモリ:8Gb(16Gb以上を推奨)
ストレージ:1Tb以上を推奨(インストール領域500Gb)
O S : Windows 10 64bit (バージョン1709以降)
サポート窓口:techsupport@w-fusion.co.jp
当社が開発してきました新型コロナウイルスのゲノム配列と型推定の技術紹介として、CoVGAS商品の紹介は当面の間公開する予定です。
今後は本技術を用い、目的に合わせたソフトウエアを受託開発を行います。
詳しくはお問い合わせください。
CoVGAS - 製品技術のご紹介
高速シーケンサデータから、新型コロナウイルス ゲノム配列・型推定を行います。
CoVGASはFluGAS同様に変異株検出に強いアルゴリズムを用いています。
コロナウイルス専用のワークフローによりシーケンサーからのデータを解析し
■ゲノム配列の決定
■参照配列に対するアミノ酸変異の検出を自動的に実施
■Nextclade 型推定、Pangolin 型推定
までを自動で実施するワークフロー(特許第6624736号)を搭載しています。本ワークフローは今後も様々なウイルスゲノム解析に有用な技術であり、当社が保有しております。

-
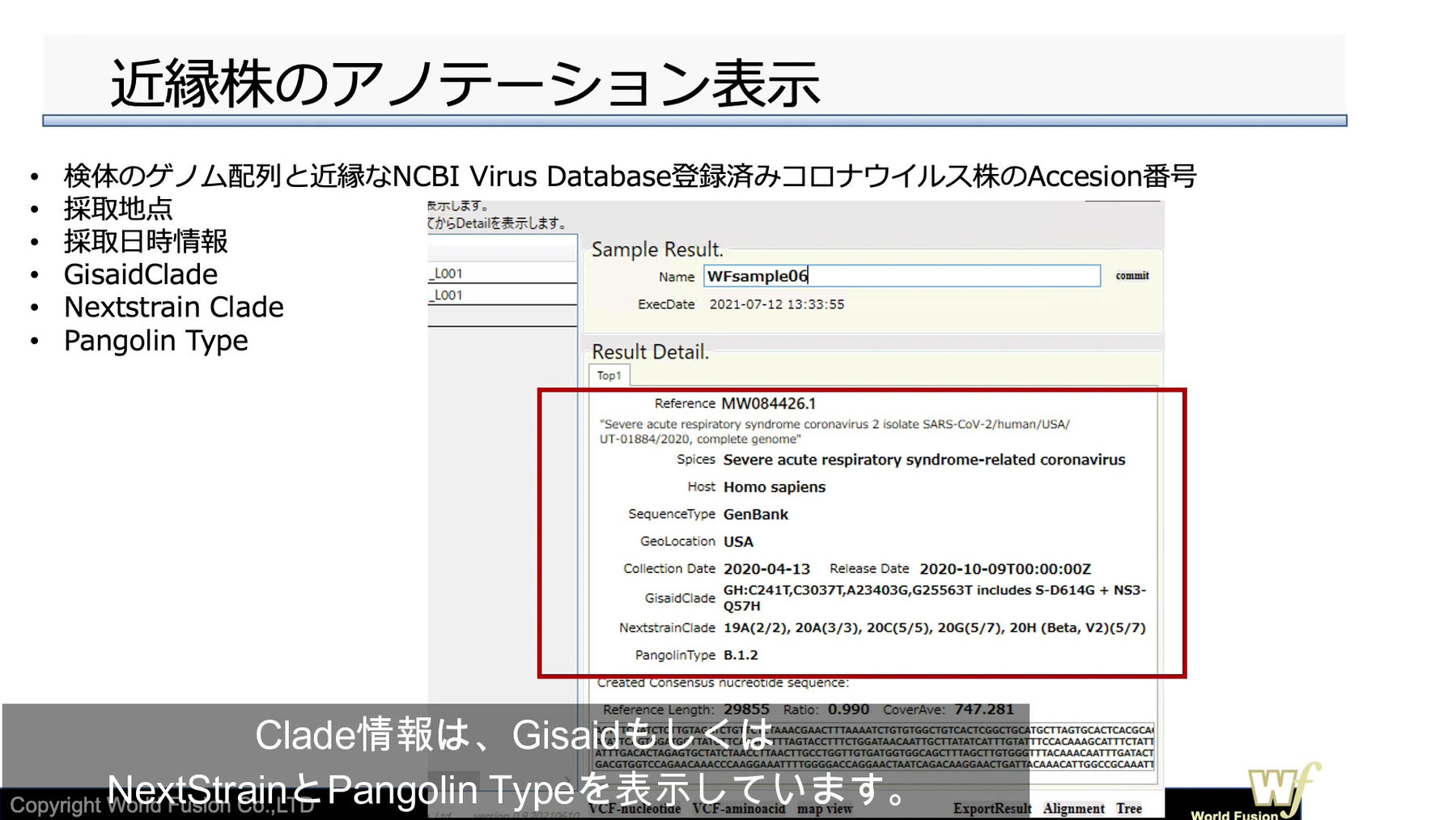
CovGASではゲノム配列解析の結果として類似株の同定、clade情報、近縁株の情報を表示します。
-
検体中に存在した新型コロナウイルスが持つゲノム配列と最も相同性が高いNCBI Virus Database登録済みのCOVID-19株に付随する情報、採取地点および採取日時情報を取得して表示します。

Windows上での解析技術と各種シーケンサーへの対応技術
-
CovGASは全てをWindows PC上で操作可能で、各シーケンスデータをそのままドラッグ&ドロップするだけで解析が開始され、自動的に結果が表示されます。
-
コマンド技術や難しい操作は不要です。シーケンサーをご利用の機関であればどこでも解析が可能となります。
-
イルミナ社MiSeqとオックスフォードナノポアシーケンサーのデータは、簡単な操作で切り替えできます。
*Nanpore シーケンサをご利用で basecall を実施せず出力したFast5ファイルの場合や、解析時に検体ごとのBarcode分割を行いたい場合はGuppy windows版をご用意いただく必要があります
CoVGAS 機能一覧
-
Fastqファイル(イルミナ社/オックスフォードナノポア社 シーケンサ)対応
-
Windows上のGUIから複数検体ファイルをドラッグアンドドロップし、ゲノム配列決定から型判定まで自動的に実行
-
検体の決定されたゲノム配列を結果画面に表示、Fasta形式にて取得可能
-
コンセンサス配列と共にNCBIに登録されている近似株の株付属情報(採取地点・日時・GenBank Accession)等の表示
-
最終コンセンサスと類似株のマッピング結果のリファレンスカバー率・平均カバレッジの表示
-
コロナ標準株へのマッピング結果より変異箇所をVCF形式で出力、アミノ酸変異箇所へのコンバート
-
アライメントビューアー(AliView), マッピングビュアー(Tablet)との連携
-
Illumina Pairend シーケンスのPrimer配列除去(Alt_nCov2019_primers)
-
検体の変異箇所からNextclade 型推定(詳細はNextclade サイトへ出力されたコンセンサス配列を入力)
-
コンセンサス配列からPangolin 型推定(Pangolin lineage)
-
最新似株配列データベース自動更新
※ Nanopore社が提供するGuppyをインストールする事により Fast5 raw-data 形式の検体データに対応します
*当ソフトウェアに組み込まれたウイルスゲノム配列解析ワークフローは特許第6624736号に登録された方法を元に作成されています。
CoVGAS紹介ビデオ
*変異検出に使用される参照配列はNC_045512(Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1)を使用。*ゲノム配列はNCBI Virusに登録されたCOVID19参照配列データベースから最適なウイルス株を参照配列と選択して実行されたマッピング結果をもとに作成。
ワークフロー型ソフトウエアの開発
CovGASの解析ワークフローの例ですが、このようなワークフロー型専用ソフトやシステムの開発を承ります。
1. シーケンスデータ
-
Fastqをドラッグアンドドロップ (複数選択可能)

2. QCチェック(自動実行)
Read dataのQuality Control (Phread scoreとRead長に基づくトリミング)の実施
3. マッピング(自動実行)
QCを通過した Fastqファイルを対象にNCBIより取得したリファレンス配列へマッピングを行います。
マッピングは、1stマッピング後コンセンサス配列を作成後、そのコンセンサス配列をもとに2ndマッピングを実施しています。本アルゴリズム詳細はお問い合わせ下さい。
4. ゲノム配列決定・類似株決定(自動実行)
上記マッピング手法で求めた方法で求めたゲノム配列をもとに類似株情報を求めます。(ウイルスにより異なります)
5. レポート
ご要望に応じたレポートの情報提供
-
この例ではNCBI に登録されているMW084426.1が最近似株としています。
-
MW084426.1の情報からサンプル取得日、取得場所などの情報を表示するしています。
-
GISAID に記載されているGH型と推定しており、Nextclade 20H (Beta, V2) 変異株にある7個の変異箇所のうち5つが共通していることを示し、Pangolin Lineage B.1.2 がアサインされています。
-
MW084426.1をリファレンスとしたマッピング結果より、平均カバレッジ、カバー率と、コンセンサス配列が表示されます。
-
Nextclade、Pangolin の文字列からブラウザが起動し、表示されているコンセンサス配列を入力することで、各サイトにおける詳細情報を得ることが可能です。
